玩转单细胞(12):单细胞celltype颜色、顺序设置及V5小问题
- 生活百科
- 2024-04-05
- 338
❞玩转单细胞往期精彩系列:玩转单细胞(2):批量做图修饰玩转单细胞(3):堆叠柱状图添加比例
玩转单细胞(4):单细胞相关性
玩转单细胞(5):单细胞UMAP图只标记特定细胞群、圈定细胞群及坐标轴修改
玩转单细胞(6):单细胞差异基因展示之对角散点图
玩转单细胞(7):修改对象基因名称
玩转单细胞(8): 单细胞3维聚类图展示
玩转单细胞(9):单细胞对象数据操作玩转单细胞(10):替换单细胞对象UMAP坐标
玩转单细胞(11):单细胞基因表达图分面设置
今天我们这一期要说的内容其实很简单,之前我们在写其他内容的时候或多或少的提到过,但是奈何问的人实在很多,次数也很多,所以索性这里出个帖子,将这个问题直接综合起来讲一下。这个内容就是我们用作图的时候,例如做降维图的时候,如何指定的颜色。用或者作图的时候如何设置顺序。那么最后还有一个小问题就是 V5 的使用,其实的更新并不是很可怕,遇到那里有错,解决就可以了!
一、指定降维图中的颜色
我们使用做将降维图的时候,每个或者的颜色是利用cols参数设置的,那么很多小伙伴就提出问题,我修改颜色的时候如何指定呢。很简单:
#==========================================================================# 1、cluster/celltype指定颜色设置#==========================================================================library(Seurat)#指定cluster/celltype的颜色Idents(uterus) <- "seurat_clusters"clusterCols <- c("#843C39", "#8C6D31", "#E6550D", "#3182BD", "#54990F","#BD9E39", "#E7BA52", "#31A354", "#E41A1C", "#6BAED6","#9ECAE1", "#AD494A", "#A1D99B", "#C7E9C0", "#99600F","#C3BC3F", "#D6616B", "#FF7F00", "#1B9E77", "#FDAE6B","#66A61E", "#F1788D", "#E6550D", "#E7969C")names(clusterCols) <- c(0:24)DimPlot(uterus, group.by='seurat_clusters', cols=clusterCols, pt.size=1, raster=F, label = T)+NoLegend()unique(uterus$celltype)# [1] "Smooth muscle cells" "Lymphocytes"# [3] "Unciliated epithelial cells" "Stromal fibroblasts"# [5] "Ciliated epithelial cells" "Endothelial cells"# [7] "Macrophages"Idents(uterus) <- "celltype"cols <- c("#843C39", "#E6550D", "#3182BD", "#54990F", "#FF7F00", "#1B9E77", "#FDAE6B")#将需要的颜色与cell type对应names(cols) <- c("Lymphocytes","Stromal fibroblasts","Unciliated epithelial cells","Endothelial cells","Smooth muscle cells","Ciliated epithelial cells","Macrophages" )DimPlot(uterus,group.by='celltype',cols=cols,pt.size=1,raster=F,label=T)+NoLegend()
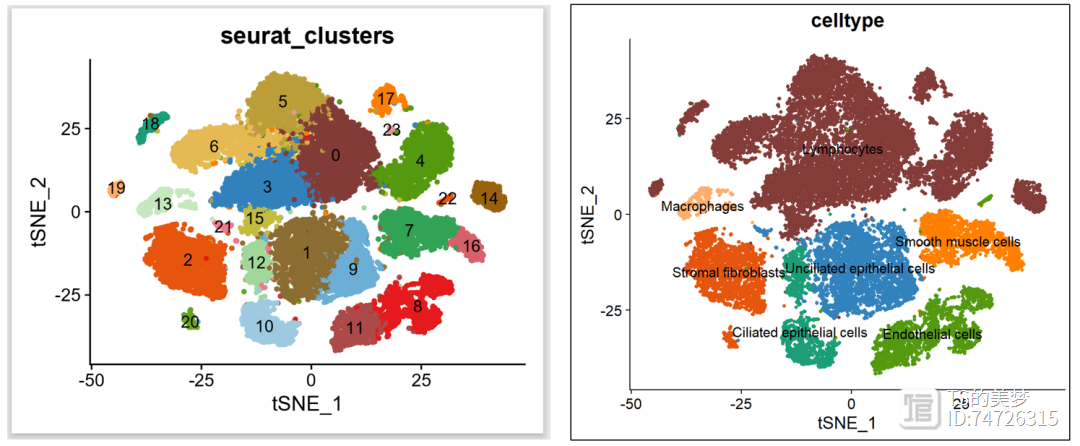
二、作图顺序的指定这个问题真的说过无数次了,设置顺序其实就是就可以了!
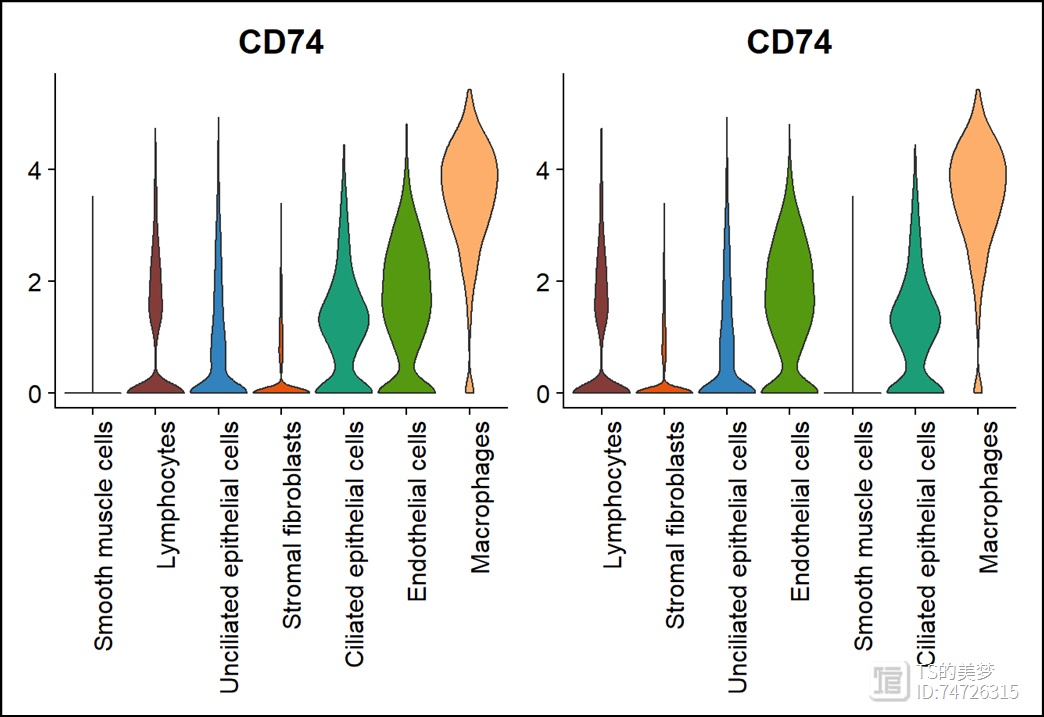
#==========================================================================# 2、seurat作图修改celltype或者cluster顺序#==========================================================================library(ggplot2)#比如我做一个小提琴图p1 = VlnPlot(uterus, features = "CD74", pt.size = 0, cols = cols)+theme(axis.title = element_blank(),axis.text.x = element_text(angle = 90))+NoLegend()Idents(uterus) <- factor(Idents(uterus), levels = c("Lymphocytes","Stromal fibroblasts","Unciliated epithelial cells","Endothelial cells","Smooth muscle cells","Ciliated epithelial cells","Macrophages" ) )p2 = VlnPlot(uterus, features = "CD74", pt.size = 0, cols = cols)+theme(axis.title = element_blank(),axis.text.x = element_text(angle = 90))+NoLegend()p1|p2
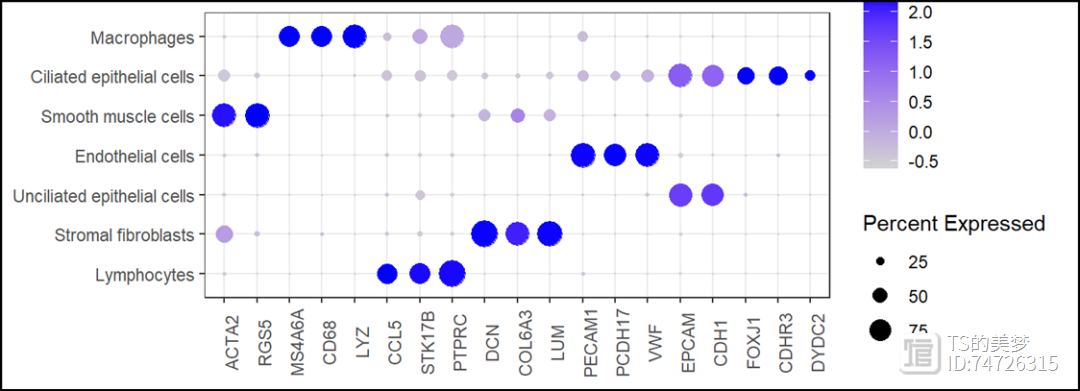
#同样的设置后,Dotplot也会随着自定义顺序显示markers <- c("ACTA2", "RGS5", #smooth muscle cells---7, 16"MS4A6A", "CD68","LYZ",#macrophages---13"CCL5", "STK17B","PTPRC",#lymphocytes---0,3,4,5,6,14,15,17,23,18,19"DCN", "COL6A3", "LUM",#stromal fibroblasts---2,20"PECAM1","PCDH17", "VWF",#endothelial cells---8,11,22"EPCAM", "CDH1",#(unciliated)epithelial cells---1,9,21"FOXJ1","CDHR3","DYDC2")#(ciliated)epithelial cells---10,12DotPlot(uterus, features = markers)+theme_bw()+theme(axis.title = element_blank(),axis.text.x = element_text(angle = 90))
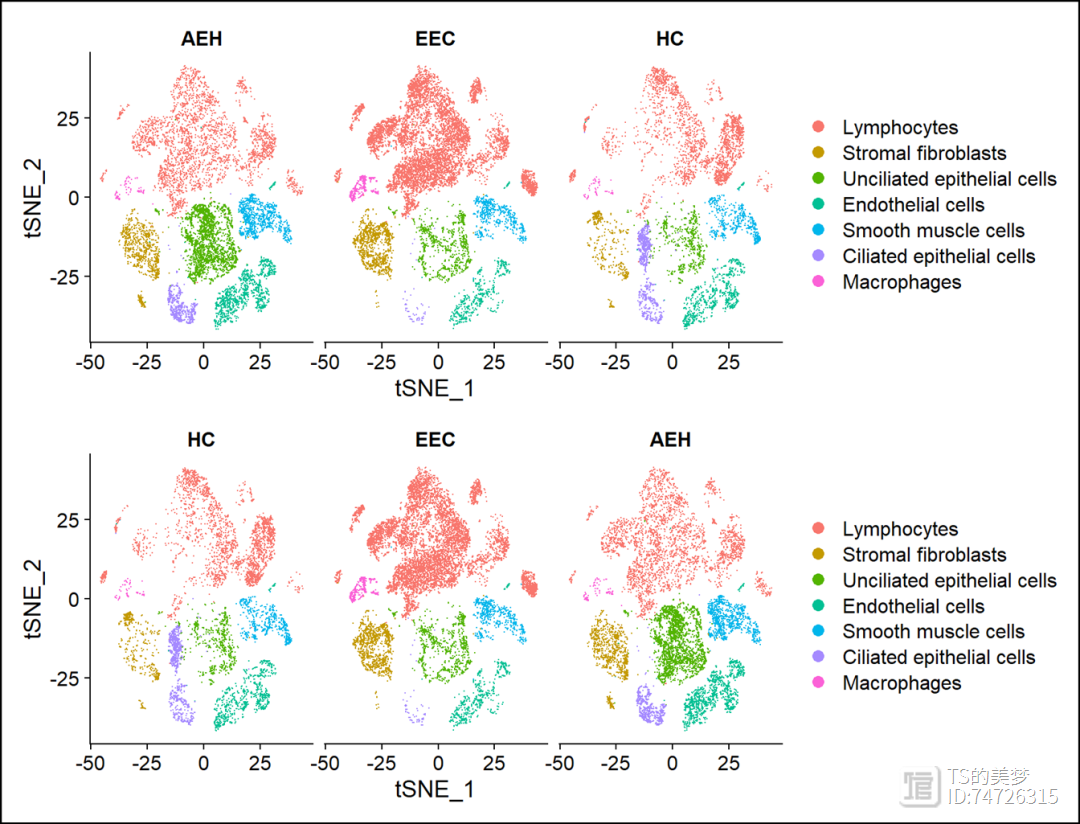
#对于split的p3 = DimPlot(uterus, split.by = "orig.ident")#可以看到celltype legend的排列是按照我们设置的顺序。但是我们需要设置分组的顺序uterus$orig.ident <- factor(uterus$orig.ident, levels = c("HC","EEC","AEH"))p4 = DimPlot(uterus, split.by = "orig.ident")p3/p4
三、 V5
更新到V5,也许你用的V4,或者不小心更新到V5,可能在使用一些内容的时候出错。或者你用的V5构建的对象,但是用之前的一些包或者教程的时候出错!不过不是什么大毛病。其实就是数据结构发生了变化,如果你关心你就取看看官网.如果不关心,那也没关系,遇到了解决就可以了这里我们以为例, obj用的V5构建的!
#==========================================================================# 3、seurat V4/V5的一个小问题示例#==========================================================================#seurat更新到V5,也许你用的V4,或者不小心更新到V5,可能在使用一些内容的时候出错。#或者你用的V5构建的seurat对象,但是用之前的一些包或者教程的时候出错!#不过不是什么大毛病。其实就是数据结构发生了变化,如果你关心你就取看看官网#如果不关心,那也没关系,遇到了解决就可以了#这里我们以scmetabolism为例,seurat obj用的V5构建的library(scMetabolism)human_countexp_Seurat<-sc.metabolism.Seurat(obj = obj,method = "AUCell",imputation =F,ncores = 2,metabolism.type = "KEGG")#很快就会出现如下的报错# Error in sc.metabolism.Seurat(obj = obj, method = "AUCell", imputation = F, :# no slot of name "counts" for this object of class "Assay5"#我们看看这个R包的原函数,我这里截图一部分:```sc.metabolism.Seurat <- function(obj, method = "VISION",imputation = F, ncores = 2,metabolism.type = "KEGG") {countexp<-obj@assays$RNA@countscountexp<-data.frame(as.matrix(countexp))#signatures_KEGG_metab <- "./data/KEGG_metabolism_nc.gmt"#signatures_REACTOME_metab <- "./data/REACTOME_metabolism.gmt"signatures_KEGG_metab <- system.file("data", "KEGG_metabolism_nc.gmt", package = "scMetabolism")signatures_REACTOME_metab <- system.file("data", "REACTOME_metabolism.gmt", package = "scMetabolism")...}```#可以看到,作者在提取count exp的时候,使用的是countexp<-obj@assays$RNA@counts#这是因为数据结构改变了。所以我们只需要修改一下函数的这里就可以了。当然了,后续如果作者更新R包那就最好了#获取矩阵使用getassaydatacountexp<-GetAssayData(obj, assay = 'RNA', layer = 'counts')# countexp<-GetAssayData(obj, assay = 'SCT', layer = 'counts')
这样就完事了,问题不大。都是一些小问题,但是很实用。觉得分享有用的,点个赞再走呗!
发表评论